FIE 针对Nature Chemistry文章的评述:利用废塑料生产具有经济吸引力的商业级汽油
(原标题:张成、钱珑联合研究团队与合作者在《自然》发文,首次提出基于并行写入策略的DNA存储方法)
团队在实验中,将中国汉代“白虎”瓦当和国宝大熊猫■◆■◆■“飞云”的高清图片成功写入DNA分子中★◆◆★★,数据量超过27.5万比特◆◆■■★■,相比此前发表的其他非传统DNA存储技术,数据规模提升超过300倍。信息读取使用便携式纳米孔测序仪,实现了对DNA模板上复杂表观比特信息的高通量读取,并通过单次超240种不同修饰模式的并行解析★◆★■■,无损还原了原始数据■★。实验结果验证了该创新型分子存储技术的可行性和准确性■★★■,还展示了表观比特的稳定性。
特别声明◆★★■★:本文转载仅仅是出于传播信息的需要,并不意味着代表本网站观点或证实其内容的真实性;如其他媒体、网站或个人从本网站转载使用◆◆★,须保留本网站注明的“来源■★◆★◆”,并自负版权等法律责任;作者如果不希望被转载或者联系转载稿费等事宜★◆★,请与我们接洽。
图灵奖得主John E. Hopcroft&北京大学郭耀教授:解读信息时代的计算机科学课程和教学
表观比特DNA存储框架为大规模数据存储提供了全新的解决方案,有望突破DNA存储的成本和速度壁垒◆◆■★。该技术的开发■◆◆★■,还展现了非传统分子比特在数据存储中的独特优势◆★◆◆■,为未来新型分子信息处理系统的研发奠定了基础。张成、钱珑、北京大学教授欧阳颀和美国亚利桑那州立大学教授颜颢为本文的共同通讯作者◆◆◆★★。吴燃峰、孙法家和林艺生为本文共一作者■◆。北京大学计算机学院和定量生物中心为本文第一通讯单位。本研究获得了德国斯图加特大学刘娜团队◆◆■★、成都瀚辰光翼科技有限责任公司、大连理工大学张强团队、华北电力大学杨静团队的大力支持。本研究的iDNAdrive实验获得了北京大学2024年iGEM团队的大力支持。该工作得到了国家重点研发计划★◆、国家自然科学基金、军委装备研究项目、北京大学-鲲鹏昇腾科教创新卓越中心项目的资助。
2024年10月23日★◆◆,北京大学计算机学院张成与定量生物学中心钱珑联合研究团队与合作者,在国际学术期刊Nature上发表题为■■★★“Parallel molecular data storage by printing epigenetic bits on DNA” 的研究论文★★■■◆★,首次提出了一种基于并行写入策略的DNA存储方法。该技术不依赖于主流的★■■★“从头合成■★◆■★”写入路线原理,通过DNA自组装与选择性酶促甲基化的组合原理,可将“表观比特”(epi-bit,5-甲基胞嘧啶)编码的数字信息并行打印在DNA分子上。同时,研究中还功实现了个人订制DNA存储示例★■■,证明了便捷的分布式DNA存储应用潜力★■■。该方法的建立◆■◆★,不仅为实现了快速、低成本的大规模分子数据存储奠定了技术基础★◆◆,还为未来DNA存储的发展提供了全新思路。
大数据时代,全球数据洪流对数据存储技术提出了严峻挑战。DNA分子具有超高的数据存储密度和超长寿命,已成为备受瞩目的颠覆性存储介质。然而,传统DNA存储依赖“从头合成”的信息写入路线◆■◆★,在成本和速度上面临巨大挑战★◆。不同于传统技术路线★■◆,张成■◆■◆、钱珑联合团队开发的“表观比特(epi-bit)”DNA存储利用预制的DNA模板和分子活字块■◆,通过DNA自组装介导的分子信息排版,经选择性酶促甲基修饰转移■★,实现了分子级“活字印刷◆◆★■◆◆”信息打印。
值得关注的是,团队还展示了这项技术的分布式存储应用潜力。在个人定制DNA存储实验中■★,邀请了北京大学、华北电力大学等单位60名背景广泛的青年志愿者★◆■★■★,由他们在日常环境下◆★★■■,将私人数据亲手写入DNA并由个人保存■■◆★◆,相关数据直到使用时才被读取★■■■,可有效保障个人数据的隐私与安全■■■。这种分布式DNA存储方式,不仅能极大降低DNA存储的使用门槛,且保障了数据隐私■★★◆◆,有望推动DNA存储的个人应用。

浪漫粉红火山、特色潍坊风筝展、丰台版的“王婆说媒”、露营狂欢、花海世界……5月18日至5月26日,2024北京丰台首届粉色熔岩浪漫纪将在丰台区王佐镇

臭豆腐之所以“闻起来臭,吃起来香”主要是因为?这是2024年蚂蚁庄园5月13日今日庄园小课堂的问题,下面为大家详细介绍2024年5月13日今天蚂蚁庄园课堂小

在“新标准”头盔实施的过渡阶段,未规范佩戴符合相关质量标准安全头盔,并系扣牢固的,交警将根据相关法律规定,处警告或30元罚款。同时督促并提醒骑乘人员

他们正在与相关机构密切合作,确保问题得到快速而有效的解决。公司强调,将会通过合法合规的方式妥善处理所有问题,并及时向公众通报处理进展,以恢复

阿卫生部称,8月中旬,在该省一医院发现1例不明原因肺炎,感染者双侧肺部存在炎症,阿卫生部正在调查其病因和传播路径,不确定是病毒还是细菌导致发病,并称
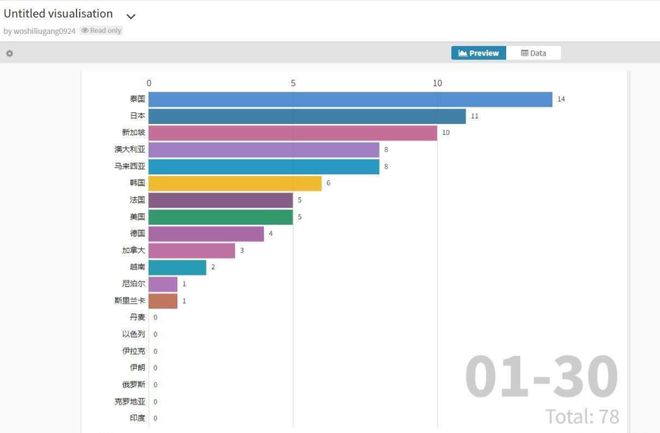
介绍:Echarts是百度推出的一个纯JavaScript 的开源可视化图表库,使用者只需要引用封装好的JS,就可以展示出绚丽的图表欢迎来到公海赌船
特别声明:以上内容(如有图片或视频亦包括在内)为自媒体平台“网易号”用户上传并发布,本平台仅提供信息存储服务。 苹果将于2028年推出OL
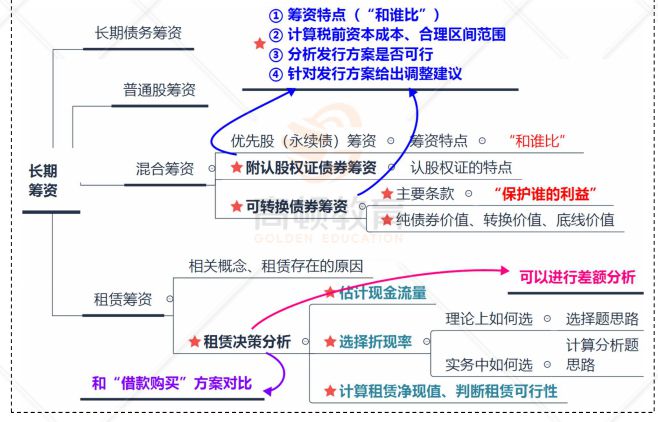
财务报表分析模板包含这套模板囊括三大财务报表,自带公式输入数据即可自动生成一键分析,高效快捷!是一个非常好的工具,针对问题有依有据,预览如下

政务公开重点领域信息公开专栏乡村振兴信息公开乡村振兴工作动态 10000 && ss.indexOf(cc.charAt(i)) != -
同时,全球湾区豪宅屹立资本顶端,独占CBD城市资源与滨水自然资源,成为引领全球房价走向的风向标。 当然,比起未来前景,我觉得更让人觉得惊喜